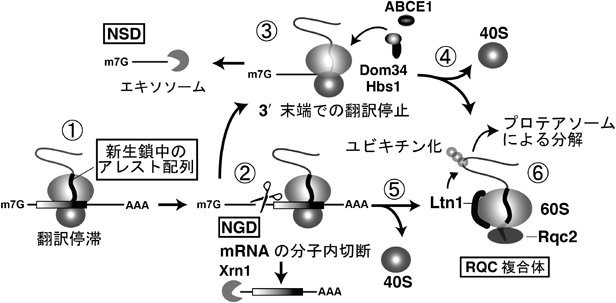
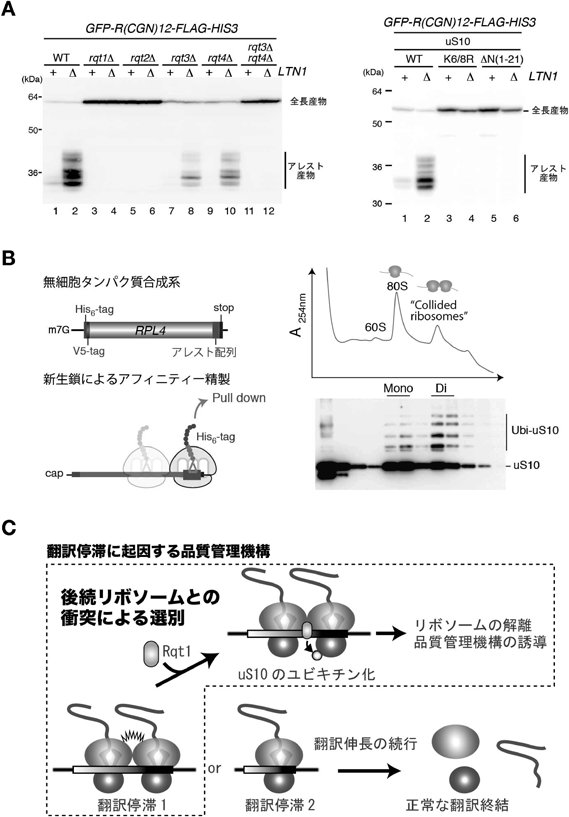
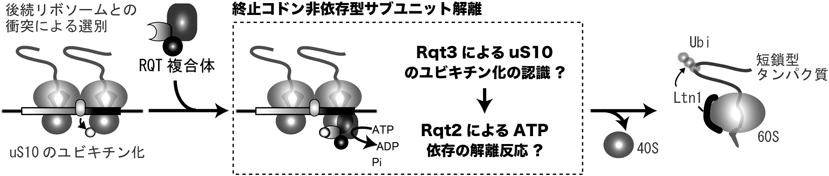
リボソームのユビキチン化による翻訳の強制終了が新生ペプチド鎖の分解を誘導するRibosome ubiquitination triggers co-translational quality control
東北大学大学院薬学研究科遺伝子制御薬学分野Graduate School of Pharmaceutical Sciences, Tohoku University ◇ 〒980–8578 宮城県仙台市青葉区荒巻字青葉6–3 C305 ◇ 6–3 Aoba, Aoba-Ku, Sendai 980–8578 Japan