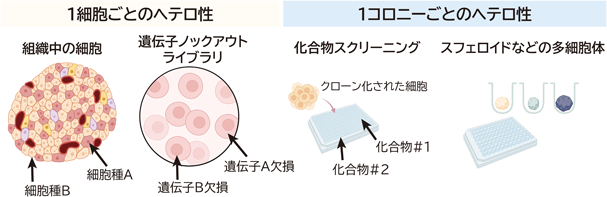
細胞集団は,しばしば不均一(ヘテロ)である.組織中の細胞はもとより,クローン細胞株のような一見均一だと思われる細胞集団でも,個々の細胞の状態や応答にはヘテロ性がある.この事実は,トランスクリプトーム解析を筆頭に,オープンクロマチン解析やイメージング解析など,多様な1細胞解析により明らかになってきた1, 2).疾患をもたらす細胞集団でも同様で,個体間,さらには,個体内の疾患細胞もヘテロであることが明らかになっている3, 4).がんオルガノイドのようなin vitroの培養系でも,同様のヘテロ性が観察される.
大量のヘテロな細胞の解析は,人工的に摂動を与える実験系でも重要となる.一つの想像しやすい例は,遺伝子ノックダウン,ノックアウトスクリーニングであろう.多様な遺伝子改変ツールの開発により,siRNAによるノックダウンや,CRISPRによるノックアウトなどを利用し,遺伝子発現パターンが多様な細胞集団を比較的容易に作製できるようになった5).そのような細胞集団の中から目的の表現型を示す細胞を取捨選択することで,特定の細胞応答に重要な遺伝子の同定が可能となる.たとえば,ノックアウト細胞ライブラリに対して細胞死誘導刺激を施し,生き残った集団の中でノックアウトされている遺伝子を同定することで,細胞死誘導に必要な遺伝子を特定できる6).この例のように,細胞集団の中の1細胞ごとの違いに注目した解析だけではなく,異なる性質を持つコロニーを大量に解析する手法もあり,その代表例が,化合物スクリーニングである(図1).均質な細胞集団をウェルなどの区画に分配し,さまざまな化合物を個々に処置した上で,細胞応答を計測する.この過程により,目的の細胞応答を誘導する化合物を同定可能となり,また,化合物の標的因子の情報から,細胞応答を制御する因子の同定につながる.実際に,創薬のプロセスでも多用されており,疾患モデル細胞などに対して化合物スクリーニングを実施することで,治療薬候補が同定されている7).
このように,細胞集団内のヘテロな1細胞や1コロニーごとの特性を個々に計測することは,生物学,医学,薬学など多面的な価値を生み出す.そのため,膨大な数の1細胞や1コロニーを,多角的な側面から解析する技術の重要性は疑う余地がない.
1)ウェルを用いた微小区画化
一般的には,市販の96ウェル,384ウェル,1536ウェルプレートなどを用いた方法が汎用されている.384ウェル,1536ウェルプレートを使用する場合,各ウェルのサイズが小さくなるため,自動分注ワークステーションがあれば,比較的ハンドリングが容易となる.それぞれのウェルは物理的に隔離されており,サンプル間の培地等の混入がないため,実験条件と実験結果が1対1で容易に紐づくことが大きな利点となる.たとえば,アレイ型CRISPRスクリーンの場合,特定の1遺伝子がノックアウトされた細胞コロニーが各ウェルに配置されるため,計測結果と遺伝子情報との紐づけが容易となる.なお,アレイ型のレンチウイルスgRNAライブラリなども市販されており,実際にスクリーニングを実施するための材料は整っている.また,化合物スクリーニングにおいても,1化合物のみを1ウェルに添加することで,各化合物の純粋な効果が計測可能となる.実際に,アカデミックな創薬支援機構や試薬ベンダーから,ウェル型の化合物ライブラリが提供されており,さまざまなスクリーニングが実施されている12, 13).計測の観点では,プレートリーダーを活用することで,細胞集団を対象としたルシフェラーゼアッセイやELISAが容易に行える.加えて,明視野イメージングと発光計測の組合わせなど,計測の多重化も比較的実施しやすい.このように,市販タイプのウェルプレートを用いた解析が,最も簡便で,さまざまな用途で広く利用されている.
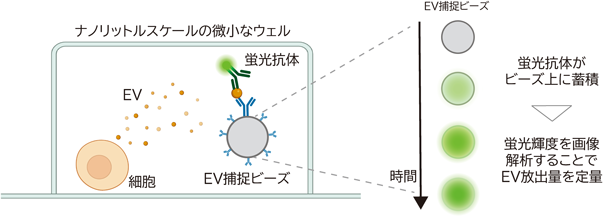
フォトリソグラフィーを用いて作製可能なマイクロウェルデバイスを使う方法もある.市販ウェルプレートがマイクロリットルスケールの反応系であるのに対して,ナノリットルスケールの,より微小なマイクロウェルを使うことで,1細胞由来の微量な液性因子の大量・並列計測などが可能となる.このような微小ウェルにおいては,微量なものを濃縮して計測可能であるというメリットがあり,ここでは,EVの1細胞計測の例をいくつかあげる.基本的な戦略は,微小な空間で1細胞を培養し,放出されたEVを抗体で捕捉し,蛍光ラベルした異なる抗体を新たに加えて定量するものである(図3).具体的には,一次元バーコード型に配置された複数の抗体アレイでEVを捕捉するものや14),抗体をコーティングしたビーズでEVを捕捉するものがある15–17).このほかにも,ウェル底に吸着させた抗体でEVを捕捉し,全反射蛍光顕微鏡で定量するものもある18).
しかし,ウェル型で大量解析を行うには本質的な課題がある.たとえば,10万の条件でスクリーニングを実施しようとすると,少なくとも260枚程度の384ウェルプレートが必要となり,5リットル程度の培地を消費する.また,自動分注機を用いた細胞の播種を,1枚あたり3分と仮定した場合でも,計13時間を要する.すなわち,サンプルが大量になるほど,解析に要する費用,時間が膨大になり,現実的に遂行が難しくなる.マイクロウェルを用いると上記の課題が解決されるアプリケーションもあるが,一部にとどまる.また,多くのマイクロウェル作製には,専門的な技術が必要となり,汎用化は容易ではない.このように,解析対象となる条件数が増加するにつれて,ウェル型解析系とは異なる,よりスケーラブルな手法が求められる.
2)浮遊型微小区画化ユニット
一般的に,浮遊型での培養・解析系は,ウェル型解析系に比べて大スケール化しやすい.たとえば,コラーゲンなどを材料とした球状の担体の表面や内側で細胞を培養することで,浮遊状態で細胞を大量培養する技術が広く用いられている.近年,微小区画化された浮遊型細胞培養ユニットの開発が進んでおり,ウェル型では達成が難しいスケールでの大量培養・解析が可能になってきている.大量サンプルの安定なハイスループット計測のためには,各培養ユニットがマイクロメートルスケールと小型であること,そして各ユニットが均一サイズであることが重要であり,これらの要件を満たすためにさまざまなマイクロ流体技術が活用されている.加えて,ソーティング技術と組み合わせることにより,特定の培養ユニットを高速に選別することが可能となり,マルチモーダル解析との接続性も高い.
a.油中水滴
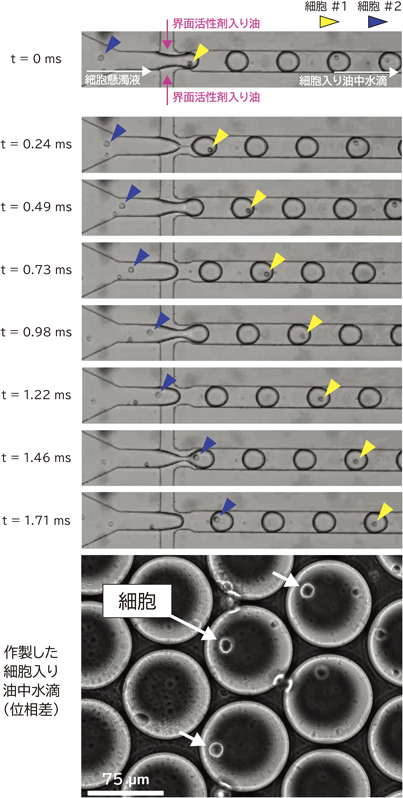
大量の微小な油中水滴が,微小区画化ユニットとして長年広く用いられており,さまざまな分子解析に活用されている.droplet digital polymerase chain reaction(ddPCR)は,DNAやRNAなどの核酸を絶対定量する高感度,高精度な計測系であり,解析のための機器も市販されている.この反応系においては,数万個の微小油中水滴中に,PCRの鋳型となる核酸が1コピー/液滴以下になるように希釈して封入し,増幅が確認された油中水滴の数を計測することで,鋳型の濃度を推定する19).シングルセルRNAシーケンシング法においても,微小油中水滴が活用されている.この手法では,油中水滴の中に1細胞と1バーコードビーズを封入することで,1細胞由来のmRNAに対して,ユニークなバーコードを付与する.油中水滴を微小区画として利用することで,各区画間のコンタミネーションを防ぎ,また,マイクロ流体技術を活用することで,その油中水滴を均一かつ高速に作製することで,高効率かつ高精度な1細胞解析が可能となる20, 21).
このように多様な分子計測に活用されている油中水滴は,細胞の培養・解析にも応用できる(図4).特に,ピコリットルスケールの空間の中に液性因子を蓄積できるため,1細胞由来の少量の液性因子を高感度に計測できるという強みがある.油中水滴内に細胞を封入するという特質上,浮遊細胞の適合度が高く,区画化された浮遊1細胞の大量解析への応用がなされている.以下に油中水滴を用いた細胞解析の例をあげる.
一つの確立されたワークフローとして,特定の抗原を認識するIgGの放出量を1細胞レベルで計測・解析する技術,DropMapがある.この手法では,B細胞一つと,抗体を捕捉するナノボディがコーティングされた磁気ビーズを複数個,一つの油中水滴内に共封入し,液滴内でサンドイッチELISAを行うことで,B細胞から放出されたIgGを定量する.ビーズの蛍光シグナルの経時計測,および,IgGの抗原タンパク質との結合度計測も可能であるため,IgG分泌の速度,特異性,そして,親和性の同時計測が達成される.背景のノイズと極力区別するため,磁気ビーズを1か所に整列させた上で蛍光計測を行うことで,検出に十分なsignal to noise ratio(SN比)を確保している22, 23).さらに,この計測技術に液滴ソーティング法とシングルセルバーコーディング法とを組み合わせたCelliGOが開発され,特異的な抗体を放出する細胞の濃縮,および,IgGの配列決定も達成されている.これらの技術群は,特異的な機能を発揮する抗体医薬の開発に大きく貢献することが期待され,企業における研究開発が進んでいる24).
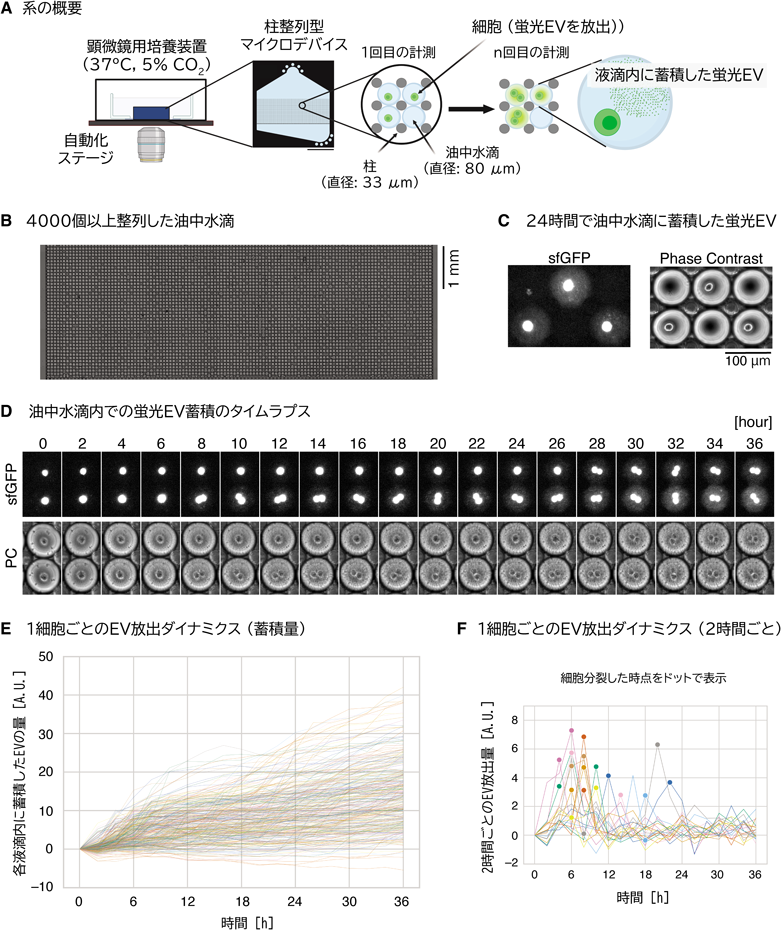
EV放出量の1細胞計測にも油中水滴系が有用である.EVマーカーに蛍光タンパク質を結合させた遺伝子を過剰発現させた細胞を油中水滴に封入し,水滴内に蓄積した蛍光EVを定量することで,1細胞由来のEV放出量の計測が可能である(図5)25).加えて,大量の液滴を整列させるマイクロデバイス中で経時計測することで,1細胞のEV放出ダイナミクスの計測が達成されている.この系を用いて初めて,細胞が分裂するタイミングでEV放出量が一時的に増加することが明らかになり,生物学的解析における1細胞計測の重要性を物語っている.
細胞間相互作用の解析にも油中水滴が活用されている.樹状細胞とT細胞との相互作用26),NK細胞と標的細胞27, 28),T細胞受容体(TCR)-T細胞と標的細胞29)との相互作用を観察した例などがあり,中でも,複数液滴の融合や特定の液滴の選別などの技術を駆使したMODEs法では,標的細胞と相互作用してIFNγを放出したCAR-T細胞の高速選別に成功している30).これら手法は,相互作用の結果を蛍光計測して,目的の細胞を選別するという戦略をとっているが,一方で,それぞれの液滴に固有のDNAバーコードを付加する手法の開発も進んでいる31).これにより,より大スケールで網羅的な細胞相互作用解析が進展することが期待される.
ここまでで紹介した細胞解析以外に,均一サイズのスフェロイドを大量に作製する際にも,油中水滴が活用されている.ピコリットルからナノリットルスケールの空間に多数の細胞を封じ込めることができるため,一般的に用いられているU底プレートなどよりも,細胞凝集を誘導しやすいものと考えられる.具体的には,主にがんスフェロイドの大量作製の実施例がある32, 33).
ここであげた例のように,油中水滴はさまざまな細胞解析や多細胞体形成に有用であることが示されてきている.一方で,培地交換が難しく,培養できる細胞種,時間に厳しい制限が加わるという課題もある.油中水滴のサイズは可変であるため,大きな水滴を用いることで1細胞あたりの培地量を増やし,細胞の生存率を上げることは可能だが,放出されたIgGやEVの濃度は下がるためこれらの計測感度は下がる.加えて,細胞間相互作用を誘導しにくくなる,細胞凝集を起こしにくくなる,などの弊害も予想される.そのため,より広範な細胞種を用いた計測や,より長時間にわたる計測などにおいては,より細胞の生存に有利な微小区画を用いた系が望まれる.
b.ハイドロゲルを用いた微小区画
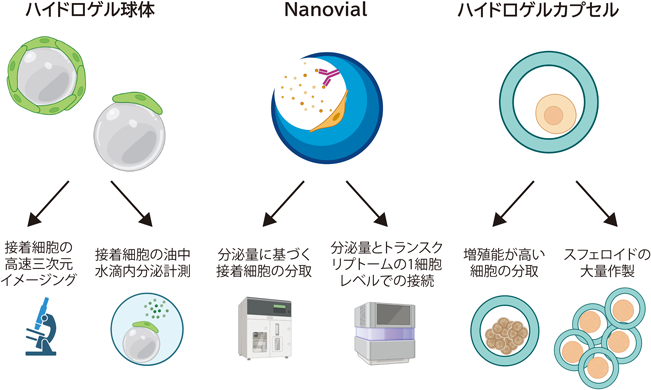
油中水滴の課題を克服するためには,水溶液で培養可能な微小区画が必要となるが,ハイドロゲルを用いた手法が有効である(図6).アガロースやアルギン酸などのハイドロゲルは,カプセルなどを形成することにより細胞を区画化することが可能でありつつも,十分な大きさの孔が空いているため,培地成分の供給が達成される.加えて,ハイドロゲルの小型培養ユニットは,市販のフローサイトメーターなどで選別することが可能であり,これまで達成が難しかった,接着した状態の細胞の選別などが可能となる.このような特性により,ハイドロゲルを用いた微小区画化は,従来の方法では困難だった細胞操作や解析を実現する有望な手法となっている.
i)ハイドロゲル球体表面での培養
直径50から100マイクロメートル程度の,均一な球状ハイドロゲルの表面で接着細胞を培養する手法がある.古くより,拡大培養のために使用されてきたマイクロスフィアと同様の培養手法であるが,マイクロ流体技術を活用することで均一なサイズの球状ハイドロゲルを作製でき,大量の均一な培養ユニットの作製が実現している.
具体的には,球状ゼラチンの表面に1細胞を接着させたものを大量に準備し,それらを油中水滴の中に封入し,大量の単一接着細胞を対象として,液性因子放出量の高速計測が達成されている34).また,ペプチド結合型アルギン酸の球状ゲル上や,ゼラチンコーティングされたマイクロキャリア上で上皮細胞の層形成も可能で,このような小型培養ユニットを用いて,接着細胞の高速三次元イメージングも実現している35).
ii)Nanovialを用いた培養
洗練された単一細胞培養・計測系として,Nanovialを用いたものがある.これは,直径数10マイクロメートルの凹みのある球体(三日月状)ハイドロゲルであり,凹みの部分にゼラチンを処置することで細胞が接着できるようにしている36, 37).マイクロ流体技術の活用で,高速かつ均質な作製が達成されており,サイズの変更に対しても柔軟である.細胞がゲル中に包埋されていないため,培地成分の供給に障壁がなく,計測のための試薬等の供給も容易である.浮遊型培養系のため,大量並列解析が可能で,市販のセルソーターでの分取も達成されている38).
この技術は,さまざまな液性因子放出の1細胞計測に応用されている.細胞が接着している凹みの部分に,液性因子に対する抗体を吸着させ,1細胞由来の液性因子を捕獲した後に,蛍光抗体でさらにラベルすることによって,液性因子の放出量定量を可能にしている39, 40).この技術を用いて,B細胞からのIgG定量,間葉系幹細胞(MSC)からのEV定量が1細胞レベルで達成されている.選別された高EV放出MSCを心筋梗塞モデルマウスに移植する実験も行われており,低放出細胞に比べて治療効果が高いことから,本手法を用いた細胞選別の医学的有用性も示されている40).
発展形として,TCRを介したT細胞の捕獲も可能になっている.主要組織適合抗原・ペプチド複合体(pMHC)をNanovialの凹みに吸着させてT細胞を捕獲した後,T細胞の活性化をIFNγの放出量で評価・選別することで,活性化したT細胞のTCRの配列決定が実現している41).この手法によって,TCRの結合能,サイトカイン放出量,および,TCRの配列情報が1細胞レベルで接続され,T細胞を用いたがん免疫療法への応用が期待される.
さらなる技術展開として,液性因子放出量とトランスクリープトームを1細胞レベルでつないだSEC-seqが開発された42, 43).この手法では,Nanovialに吸着させた抗体で1細胞由来のサイトカインを捕獲したのち,バーコードつき抗体を処置し,シーケンシングによりバーコードを定量することで,サイトカイン定量を可能にしている.より具体的には,シングルセルシーケンシングではなく,シングルゲルシーケンシング,すなわち,Nanovial一つに対してユニークなDNAバーコードを付加したシーケンシングを行っている.単一細胞由来のトランスクリプトームとサイトカインに結合した抗体のバーコードを同時に読むことで,1細胞の遺伝子発現パターンと特定のサイトカイン放出量の同時計測に成功している.
iii)ハイドロゲルカプセルを用いた培養
最後に浮遊細胞の区画化に適した,小型ハイドロゲルカプセルについて紹介する.Pico-shellは,油中水滴内でポリエチレングリコール(PEG)とデキストランを相分離させることにより作製する,直径90 µm程度の中空ハイドロゲルカプセルである.マイクロ流体デバイスを用いた高速大量作製が可能であり,藻類や酵母の培養に加え,CHO細胞の培養例が報告されている.市販のソーターを用いたpico-shellの選別も行われており,増殖能が高いクロレラの濃縮に成功している44).また,異なる種類のハイドロゲルカプセルを用いて,その内壁に上皮細胞層を形成させ,細胞が生み出す応力の解析にも活用されている45).
カプセル型構造は,スフェロイドなどの多細胞体の大量培養にも有効であり,複数の報告がある.スフェロイドの作製には通常,U底ウェルプレートが用いられるが,作製可能なスフェロイドの数がウェル数に限定され,大量作製には時間とコストがかかる.1ウェルに多数のマイクロウェルが配置された商品も市販されており,プレート1枚で,数万個のスフェロイドの作製が可能になっているものの,ウェル内で浮遊したスフェロイドどうしの融合など,大量・均質作製への課題は残る.そこで,均一サイズのカプセルを大量に作製できるというマイクロ流体技術の利点を活かし,さまざまなスフェロイドを大量かつ均質に作製する手法が開発されてきた46–49).これにより,これまで達成が難しかった,再現性の高い三次元細胞体を用いたスクリーニング系の開発が進むものと考えられる.